业务咨询

![]() 发布时间:2024-01-12
发布时间:2024-01-12
![]() 环特生物
环特生物
![]() 浏览次数:3214
浏览次数:3214

编者按
今天,我们特别关注一项于2021年2月发表在《Nature Communications》的新研究——《Genomic aberrations after short-term exposure to colibactin-producing E. coli transform primary colon epithelial cells》,该研究开发了来源于正常小鼠结肠上皮细胞的原代细胞培养模型,利用pks+大肠杆菌或大肠杆菌素合成缺陷的等基因突变体感染类器官和源自原代结肠上皮细胞的极化单层细胞,重现了感染的早期反应。
论文翻译:何玲玲
01、研究背景
结直肠癌(CRC)是全球癌症相关死亡的第二大原因。驱动癌变的基因突变已被很好地探索,例如Wnt、KRAS、TGF-β和p53通路[1]。这些通路中的突变组合负责上皮细胞的转化[2,3],并且活跃的Wnt信号被认为是几乎所有CRC的关键特征[4]。Wnt信号驱动结肠细胞增殖,仅在干细胞腔室中具有生理活性,而分化的细胞缺乏Wnt信号[5]。实验和来自患者的数据表明,组成性活跃Wnt信号是CRC级联的早期事件,通常通过APC突变和随后的杂合性(LOH)丢失来实现[6]。最近的数据同样表明,环境因素是CRC的重要驱动因素,结肠微生物群在CRC[7,8,9]的发生和进展中都起着重要作用,某些定殖于肠道的细菌似乎通过基因毒素直接影响上皮细胞的基因组完整性。
大肠杆菌素是由pks致病性岛编码的基因毒素,由几种肠杆菌科成员产生,例如属于系统发育类B2的大肠杆菌。pks+大肠杆菌感染真核细胞系可引起DNA双链断裂(DSBs),导致巨噬细胞增多和细胞周期阻滞[10]。在炎症性肠病(IBD)、家族性腺瘤性息肉病(FAP)和CRC患者中,已经观察到携带pks基因的结肠粘膜相关大肠杆菌的增加[11,12,13]。大肠杆菌素通过共价修饰使DNA烷基化,并在培养的哺乳动物细胞和体内形成稳定的腺嘌呤-大肠杆菌素复合物[14,15,16],从而在结直肠癌患者中留下特定的突变特征[17,18]。此外,在不同的FAP和CRC小鼠模型中,pks+大肠杆菌促进了肿瘤的发生[19]。值得注意的是,在这些小鼠模型中,肿瘤也可以在缺乏大肠杆菌素的情况下出现,这就不确定大肠杆菌素是否只是促进了肿瘤的发生,或者它是否也起到了致病作用。
目前为了研究pks+大肠杆菌在体外引起的基因组不稳定性(CIN),只对转化或永生化的细胞系进行了感染,尚不清楚健康的原代结肠上皮细胞是否以同样的方式对感染作出反应。为了解决这一问题,该研究团队开发了来源于正常小鼠结肠上皮细胞的原代细胞培养模型。其利用pks+大肠杆菌或大肠杆菌素合成缺陷的等基因突变体感染类器官和源自原代结肠上皮细胞的极化单层细胞,重现了感染的早期反应。
02、主要研究成果
1、在原代结肠上皮细胞培养中,pks+大肠杆菌引起DNA损伤、巨噬细胞增多和多核细胞的形成
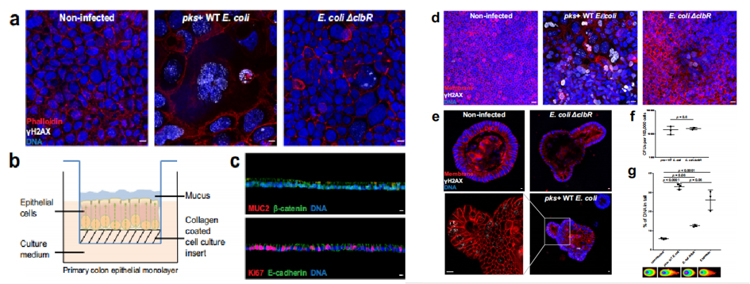
研究人员成功构建了在气液界面中生长的极化上皮单层细胞(图1b、1c),利用大肠杆菌素(pks+)野生型(WT) M1/5大肠杆菌感染该细胞3小时。为了确保大肠杆菌素具有特异性,研究人员使用大肠杆菌ΔclbR突变株作为阴性对照,该菌株对大肠杆菌素合成有缺陷。在MOI值为5的浓度下感染3小时下可检测到γH2AX(DNA损伤修复蛋白)阳性细胞、巨噬细胞增多和多核细胞(图1d),这些表型在感染ΔclbR突变体后未见,与Caco-2细胞系的表型相似(图1a)。
研究人员进一步使用pks+大肠杆菌在MOI 5下感染小鼠结肠类器官3小时,类器官首先被分解,因此基部和根尖两侧都暴露在细菌中。3天后,在类器官重构后进行全载免疫荧光标记。同样地,在感染pks+ WT大肠杆菌菌株后才观察到γH2AX阳性细胞,这表明该系统适合研究大肠杆菌素对原代细胞的影响(图1e)。
为了证实在原代细胞感染情况下观察到的影响不是由于WT和ΔclbR突变型大肠杆菌之间的感染性差异,我们对类器官感染进行了集落形成单位(CFU)分析,证实了相同的定植率(图1f)。为了进一步证实DSBs的存在,研究人员用中性彗星法测量了小鼠结肠类器官细胞中受损的DNA。结果显示,与未感染的情况相比,pks+ WT大肠杆菌感染造成了更多的DNA损伤(图1g)。
原文图1
2、pks+大肠杆菌感染后类器官的wnt非依赖性生长
为进一步探索DNA修复机制下的非永生化原代细胞的反应,研究人员用pks+大肠杆菌感染初代极化上皮单层,免疫标记γ-H2AX和Ki67。感染3 h后,有部分细胞呈现γ-H2AX和Ki67双阳性,表明它们在DNA损伤的情况下仍能增殖。这种不完全修复的DNA损伤下的细胞增殖可引起体细胞突变,这可能导致独立于生态位因子;这种生态位逃逸是癌症类器官的一个特征。
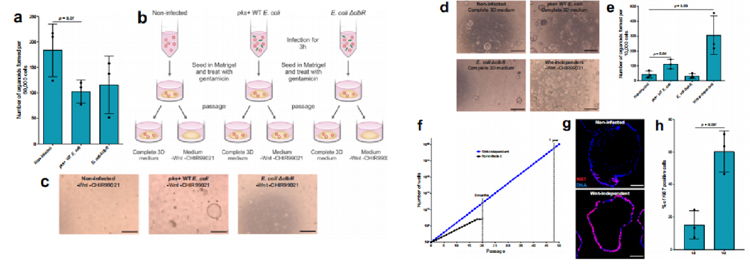
为了评估这类类器官形成能力,我们在MOI值为5的浓度下感染类器官3小时,将它们重新播种在Matrigel中,并用庆大霉素处理以杀死细菌。为了确定细胞死亡的程度,我们进行了一项类器官形成试验,在每种条件下播种50,000个细胞。我们观察到pks+ WT大肠杆菌感染的类器官形成效率降低了约40%,但WT和ΔclbR突变型大肠杆菌感染条件之间没有差异(图2a)。
由于CRC中最早和最常观察到的突变是导致Wnt通路激活的突变,在接下来的传代中,研究人员将类器官重新播种到含有Wnt和Wnt激动剂CHIR99021的完整3D培养基中,以及缺乏这两种因子的培养基中(图2b)。
结果显示,未感染和ΔclbR突变感染的类器官在无wnt培养基中不能重新生长,而在pks+ WT大肠杆菌感染的条件下,一些类器官可重新形成(图2c)。这些不依赖wnt的类器官可以进一步传代和扩张。因此,将原代细胞短时间暴露于产生大肠杆菌素的大肠杆菌中,就足以引起体细胞适应,从而产生不依赖wnt的克隆。
原文图2
3、不依赖wnt的类器官表现出增强的类器官形成能力、更高的增殖能力和寿命
为了表征感染对类器官生长的影响,研究人员测量了未感染、pks+ WT大肠杆菌感染和ΔclbR突变感染的类器官以及之前实验中产生的不依赖wnt的类器官的类器官形成能力。结果显示,与未感染或感染ΔclbR突变体的细胞相比,从WT pks+大肠杆菌感染条件下获得的细胞具有更高的类器官形成能力。然而,来自wnt非依赖性类器官的细胞容量最高(图2d, e),传代率也更高,反映了它们的生长能力增加(图2f)。因此,传代7天后的类器官免疫标记显示,与未感染的类器官相比,不依赖wnt的类器官具有更高比例的ki67阳性细胞(图2g和h)。
4、不依赖Wnt的类器官显示Wnt/β-catenin信号上调和分化基因下调
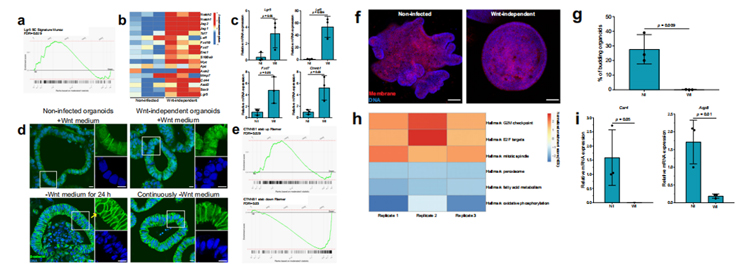
为了确定干细胞相关通路是否在不依赖wnt的类器官中上调研究人员对未感染和不依赖wnt的类器官进行了RNA测序及RT-qPCR。结果显示,与未感染的类器官相比,不依赖wnt的类器官中Lgr5特征基因显著富集(图3a)、Wnt/β-catenin相关基因上调(图3b、3c)。β-catenin免疫染色显示,无论Wnt存在与否,核β-catenin都存在于不依赖wnt的类器官中,表明Wnt/β-catenin信号通路活跃。未感染的类器官在没有Wnt的情况下保存24小时,在几个细胞核中没有细胞核β-catenin蛋白(图3d)。此外,与未感染的类器官相比,与β-catenin的稳定性相关的Wnt信号基因组在不依赖wnt的类器官中分别显示出显著的正向和负向富集(图3e),这表明不依赖wnt类器官对内源性Wnt具有更高的敏感性。
肠道类器官中Wnt信号的上调已被证明可导致细胞呈现增殖性祖细胞表型,从而导致细胞从不对称的隐窝绒毛结构(未感染的类器官)转变为球体、囊肿样形态(不依赖wnt的类器官),缺乏分化的细胞类型(图3f)。
此外,GSEA揭示了增殖相关的基因组上调、脂肪酸代谢相关的基因组下调,这些基因组在分化的结肠细胞中是活跃的(图3h);肠细胞特异性分化基因碳酸酐酶4 (Car4)和水通道蛋白8 (Aqp8)在不依赖wnt的类器官中下调(图3i)。因此,感染诱导的类器官wnt独立性伴随着增殖细胞表型和分化受损。
原文图3
5、不依赖wnt的类器官具有主要的染色体畸变和高突变负荷
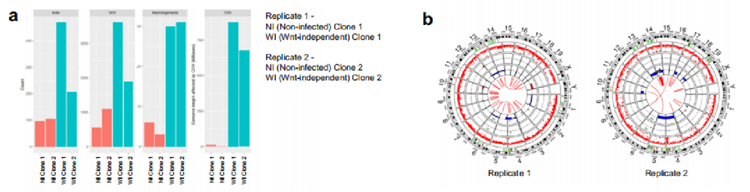
为了确定大肠杆菌素诱导的改变是否与突变有关,我们从未感染和不依赖wnt的类器官中产生克隆和混合培养物,并对两个克隆中获得的细胞进行全基因组(WGS)和全外显子组测序(WXS)。结果显示,与未感染的对照组相比,不依赖wnt类器官中存在更多的克隆(等位基因比例>25%)单核苷酸变异、插入和缺失突变,以及重排断点。在由wnt非依赖性类器官产生Replicate 1和Replicate 2中,分别约有8.75亿个和6.5亿个碱基对受到拷贝数变异(CNV)的影响(图4a)。因此,不依赖wnt的细胞表现出CNV的CIN特征,包括全染色体或染色体臂的主要损失(图4b)。
原文图4
03、编者点评
作为健康美丽产业CRO服务开拓者与引领者、斑马鱼生物技术的全球领导者,环特生物搭建了“斑马鱼、类器官、哺乳动物、人体”四位一体的综合技术服务体系,开展健康美丽CRO服务、科研服务、智慧实验室搭建三大业务。目前,环特类器官平台已成功搭建多种肿瘤及正常类器官的培养平台,欢迎有需要的读者垂询!
参考文献
1. Fearon, E. R. & Vogelstein, B. A genetic model for colorectal tumorigenesis.Cell 61, 759–767 (1990).
2. Drost, J. et al. Sequential cancer mutations in cultured human intestinal stem cells. Nature 521, 43–47 (2015).
3. Matano, M. et al. Modeling colorectal cancer using CRISPR-Cas9-mediated engineering of human intestinal organoids. Nat. Med. 21, 256–262 (2015).
4. The Cancer Genome Atlas Network Comprehensive molecular characterization of human colon and rectal cancer. Nature 487, 330–337 (2012).
5. Barker, N. et al. Identification of stem cells in small intestine and colon by marker gene Lgr5. Nature 449, 1003–1007 (2007).
6. Miyoshi, Y. et al. Somatic mutations of the APC gene in colorectal tumors:mutation cluster region in the APC gene. Hum. Mol. Genet. 1, 229–233 (1992).
7. West, N. R., McCuaig, S., Franchini, F. & Powrie, F. Emerging cytokine networks in colorectal cancer. Nat. Rev. Immunol. 15, 615–629 (2015).
8. Brentnall, T. A. et al. Mutations in the p53 gene: an early marker of neoplastic progression in ulcerative colitis. Gastroenterology 107, 369–378 (1994)
9. Fujita, M. et al. Genomic landscape of colitis-associated cancer indicates the impact of chronic inflammation and its stratification by mutations in the Wnt signaling. Oncotarget 9, 969–981 (2018).
10. Hussain, S. P. et al. Increased p53 mutation load in noncancerous colon tissue from ulcerative colitis: a cancer-prone chronic inflammatory disease. Cancer Res. 60, 3333–3337 (2000).